Aplicamos tres técnicas diferentes de optimización de hiperparámetros en un conjunto de modelos de clasificación para comparar su precisión.
Entre ellos veremos el grid, el random, y la popular optimización bayesiana!
Antecedentes
A menudo he recibido artículos que se refieren a automatizar la búsqueda óptima de hiperparámetros. Una vez que he entendido la intención y el alcance de estas técnicas, me he preguntado cuánto se puede mejorar un modelo a través de esta optimización.
El objetivo de este artículo es investigar las diferentes técnicas de optimización disponibles y probarlas en un ejemplo simple, compararlas y ver una descripción general de las mejoras obtenidas.
Los hiperparámetros son parámetros que se pueden ajustar en la creación de un modelo. Estos deben estar bien configurados para obtener un rendimiento óptimo. Entonces, optimizar los hiperparámetros de un modelo es una tarea crucial para aumentar el rendimiento del algoritmo seleccionado.
Necesitamos conocer, hasta cierto punto, la implicación que tiene cada hiperparámetro en cada algoritmo y sus posibles valores. Entender en profundidad el significado de cada uno de estos hiperparámetros en los diferentes algoritmos es algo necesario y una tarea enorme que a veces implica conocer el funcionamiento del algoritmo internamente y las matemáticas que hay detrás. El contenido de este artículo no llega a esa profundidad, aunque usaremos diferentes algoritmos a los que ajustaremos algunos hiperparámetros para analizar como varían los resultados.
Cualquiera que haya utilizado algún algoritmo probablemente ya haya realizado algunos intentos de optimización manual en el conjunto de valores predeterminado. Este ajuste manual suele llevar mucho tiempo, no siempre se realiza de forma rigurosa y dificulta la sistematización de los resultados.
En segundo lugar, podemos aplicar técnicas disponibles automatizadas y bastante sencillas como Grid Search y Random Search, que suelen dar mejores resultados, pero con un elevado coste de tiempo y recursos computacionales. Aplicaremos ambas técnicas para comparar sus resultados.
Finalmente, aplicaremos la optimización bayesiana, que es un método para encontrar el mínimo de una función, utilizando la biblioteca Hyperopt de Python con el mejor de los algoritmos probados. La implementación de esta técnica puede que no sea tan fácil, pero puede darnos mejores resultados en rendimiento o tiempo que las anteriores.
El Dataset
El conjunto de datos que usaremos para el ejercicio es el Dataset de Pasajeros del Titanic de Kaggle , que es un ejercicio de clasificación binaria para predecir qué pasajeros han sobrevivido y cuáles no.
import os
import numpy as np # linear algebra
import pandas as pd #
from datetime import datetime
from sklearn.model_selection import train_test_split
from sklearn.linear_model import SGDClassifier
from sklearn.linear_model import RidgeClassifier
from sklearn.neighbors import KNeighborsClassifier
from sklearn.svm import SVC
from sklearn.ensemble import BaggingClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.linear_model import LogisticRegression
import lightgbm as lgb
from sklearn.metrics import confusion_matrix
import scikitplot as skplt
%matplotlib inline
np.random.seed(7)
train = pd.read_csv('./data/train.csv', index_col=0)
y = train.Survived #.reset_index(drop=True)
features = train.drop(['Survived'], axis=1)
features.head()
La métrica seleccionada es acurracy¹: el porcentaje de pasajeros predicho correctamente.
Acurracy = (TP + TN) / (Total)
TP: True Positive (Positivos Verdaderos)
TN: True Negative (Negativos Verdaderos)
Feature engineering y Dataframe de resultados:
Vamos a aplicar técnicas fundamentales de la ingeniería de datos que nos permitirán utilizar los algoritmos sin errores (recuerda que no es el objetivo de este artículo obtener un buen resultado, solo comparar el rendimiento al aplicar técnicas de optimización).
Seleccionamos ocho algoritmos para realizar el trabajo; esta decisión se basa en reutilizar parte del trabajo de Jason Brownlee ² y Will Koehrsen³ que han desarrollado gran parte del código aquí utilizado.
Cada uno de estos modelos será una columna diferente en nuestra tabla de resultados:
1. Stochastic Gradient Boosting
2. Ridge Classifier
3. K-Nearest Neighbors (KNN)
4. Support Vector Machine (SVM)
5. Bagged Decision Trees
6. Random Forest
7. Logistic Regression
8. LGBM
Para cada una de estas columnas, intentaremos aplicar las siguientes técnicas de optimización:
- Hiperparámetros predeterminados
- Sklearn GridSearchCV
- Sklearn RandomizedSearchCV
- Hyperopt para Python
features = features.drop(['Cabin'], axis=1)
features = features.drop(['Name'], axis=1)
objects = [col for col in features.columns if features[col].dtype == "object"]
features.update(features[objects].fillna('None'))
numeric_dtypes = ['int16', 'int32', 'int64', 'float16', 'float32', 'float64']
numerics = [col for col in features.columns if features[col].dtype in numeric_dtypes]
features.update(features[numerics].fillna(0))
features.info()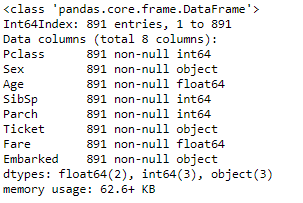
# aplicando dummies y split
X = pd.get_dummies(features)
X_train, X_valid, y_train, y_valid = train_test_split(X,y,train_size=0.70,test_size=0.30,random_state=0)
#Dataframe de resultados
cols = ['Case','SGD','Ridge','KNN','SVM','Bagging','RndForest','LogReg','LGB']
resul = pd.DataFrame(columns=cols)
resul.set_index("Case",inplace=True)
resul.loc['Standard'] = [0,0,0,0,0,0,0,0]
resul.loc['GridSearch'] = [0,0,0,0,0,0,0,0]
resul.loc['RandomSearch'] = [0,0,0,0,0,0,0,0]
resul.loc['Hyperopt'] = [0,0,0,0,0,0,0,0]
resul.head()
Fila 1: hiperparámetros predeterminados de cada algoritmo
Como se mencionó anteriormente, la primera fila de nuestra tabla de resultados es el punto de partida del análisis, tomando los valores predeterminados para los hiperparámetros de cada uno de los algoritmos:
#Creación de Modelos
sgd = SGDClassifier()
ridge = RidgeClassifier()
knn = KNeighborsClassifier()
svc = SVC(gamma='auto')
bag = BaggingClassifier()
rf = RandomForestClassifier(n_estimators=10)
lr = LogisticRegression(solver='liblinear')
lgg = lgb.LGBMClassifier()
models = [sgd,ridge,knn,svc,bag,rf,lr,lgg]
col = 0
for model in models:
model.fit(X_train,y_train.values.ravel())
resul.iloc[0,col] = model.score(X_valid,y_valid)
col += 1
resul.head()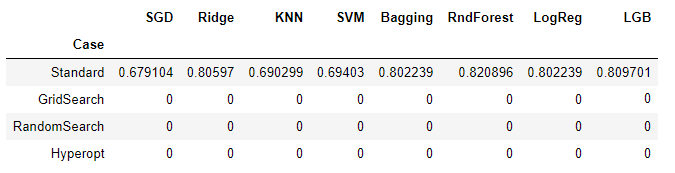
Fila 2: aplicación de GridSearchCV
“El algoritmo GridSearchCV funciona probando todas las combinaciones posibles de parámetros que se desean probar en el modelo. Cada uno de esos parámetros se prueba en una serie de iteraciones de validación cruzada (Cross Validation). Esta técnica ha estado en boga durante los últimos años como una forma de afinar los modelos ”⁴.
Ahora debemos definir un diccionario con los diferentes parámetros y sus valores para cada algoritmo
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import RepeatedStratifiedKFold
#SGD
loss = ['hinge', 'modified_huber', 'log']
penalty = ['l1','l2']
alpha= [0.0001,0.001,0.01,0.1]
l1_ratio= [0.15,0.05,.025]
max_iter = [1,5,10,100,1000,10000]
sgd_grid = dict(loss=loss,penalty=penalty,max_iter=max_iter,alpha=alpha,l1_ratio=l1_ratio)
#Ridge
alpha = [0.1, 0.2, 0.3, 0.4, 0.5, 0.6, 0.7, 0.8, 0.9, 1.0]
ridge_grid = dict(alpha=alpha)
#K-Nearest - Neighborg
n_neighbors = range(1, 21, 2)
weights = ['uniform', 'distance']
metric = ['euclidean', 'manhattan', 'minkowski']
knn_grid = dict(n_neighbors=n_neighbors,weights=weights,metric=metric)
#Support Vector Classifier
kernel = ['poly', 'rbf', 'sigmoid']
C = [50, 10, 1.0, 0.1, 0.01]
gamma = ['scale']
svc_grid = dict(kernel=kernel,C=C,gamma=gamma)
#Bagging Classifier
n_estimators = [10, 100, 1000]
bag_grid = dict(n_estimators=n_estimators)
#Random Forest
n_estimators = [10, 100, 1000,10000]
max_features = ['sqrt', 'log2']
rf_grid = dict(n_estimators=n_estimators,max_features=max_features)
#Logistic Regression
solvers = ['newton-cg', 'lbfgs', 'liblinear']
penalty = ['l2']
c_values = [100, 10, 1.0, 0.1, 0.01]
lr_grid = dict(solver=solvers,penalty=penalty,C=c_values)
#LGB
class_weight = [None,'balanced']
boosting_type = ['gbdt', 'goss', 'dart']
num_leaves = [30,50,100,150] #list(range(30, 150)),
learning_rate = list(np.logspace(np.log(0.005), np.log(0.2), base = np.exp(1), num = 10)) #1000
lgg_grid = dict(class_weight=class_weight, boosting_type=boosting_type, num_leaves=num_leaves, learning_rate =learning_rate)Luego, se aplica GridSearchCV para cada uno:
models = [sgd,ridge,knn,svc,bag,rf,lr,lgg]
grids = [sgd_grid,ridge_grid,knn_grid,svc_grid,bag_grid,rf_grid,lr_grid,lgg_grid]
col = 0
for ind in range(0,len(models)):
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3,random_state=1)
grid_search = GridSearchCV(estimator=models[col],
param_grid=grids[col], n_jobs=-1, cv=cv,
scoring='accuracy',error_score=0)
grid_clf_acc = grid_search.fit(X_train, y_train)
resul.iloc[1,col] = grid_clf_acc.score(X_valid,y_valid)
col += 1
resul.head()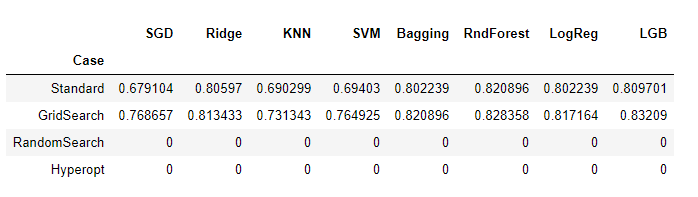
Los diferentes algoritmos tienen muchos más parámetros que no se han incluido aquí deliberadamente, y que se podrían incluir para búsquedas más extensas de valores para cada hiperparámetro.
Esta elección en el número de posibles combinaciones se hace en base al tiempo y recursos computacionales que estemos dispuestos a invertir.
Fila 3: aplicación de RandomSearchCV
“Ingrese al Randomized Search. Considere que probar todas las combinaciones posibles (de hiperparámetros) requieren mucha fuerza bruta computacional. Los científicos de datos son un grupo impaciente, por lo que adoptaron una técnica más rápida: muestrear al azar a partir de una variedad de parámetros. La idea es que se cubrirá el conjunto de parámetros casi óptimo más rápido que utilizando el GridSearch. Esta técnica, sin embargo, es ingenua (naive). No sabe ni recuerda nada de sus ejecuciones anteriores ". ⁴
A efectos prácticos, el código es idéntico a GridSearchCV modificando:
random_search = RandomizedSearchCV(models[col],
param_distributions=grids[col],n_iter=n_iter_search,
cv=cv)en vez de:
grid_search = GridSearchCV(estimator=lr, param_grid=lr_grid,
n_jobs=-1, cv=cv, scoring='accuracy',error_score=0)Aquí el código completo:
from scipy.stats import randint as sp_randint
from sklearn.model_selection import RandomizedSearchCV
col = 0
for ind in range(0,len(models)):
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3,
random_state=1)
n_iter_search = 3
random_search = RandomizedSearchCV(models[col],
param_distributions=grids[col],n_iter=n_iter_search, cv=cv)
random_search.fit(X_train,y_train)
resul.iloc[2,col] = random_search.score(X_valid,y_valid)
col += 1
resul.head()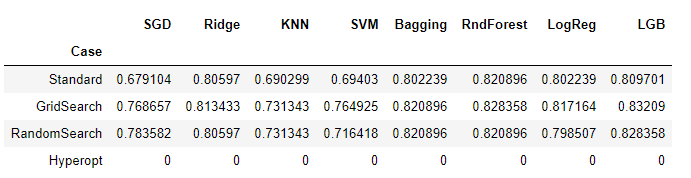
Analizando los resultados
Hasta ahora, si revisamos la cuadrícula de resultados, encontramos que la aplicación de técnicas de búsqueda automatizada ha dado buenos resultados.
En algunos casos, como el SGD o el algoritmo SVM se ha mejorado mucho, de un piso de 67-69% a un 78-76%.
La tendencia general ha sido mejorar 1, 2 o 3 puntos porcentuales, obteniendo mejores resultados con GridSearchCV que con RandomSearchCV, siendo los tiempos de aplicación de este ultimo mejores que los del Grid.
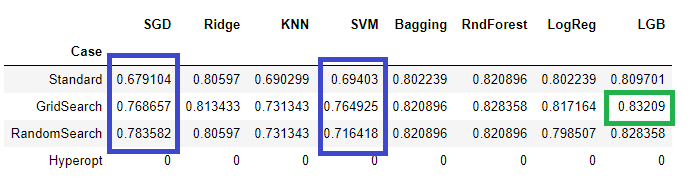
Analicemos el algoritmo ganador (Light Gradient Boost) en su versión de GridSearchCV:
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3,
random_state=1)
n_iter_search = 3
grid_search = GridSearchCV(estimator=lgg, param_grid=lgg_grid,
n_jobs=-1, cv=cv, scoring='accuracy',error_score=0)
grid_win = grid_search.fit(X_train, y_train)
#Valores predichos basados en los nuevos parametros
yv_pred = grid_win.predict(X_valid)
print(confusion_matrix(y_valid, yv_pred))
skplt.metrics.plot_confusion_matrix(y_valid, yv_pred,figsize=(8,8))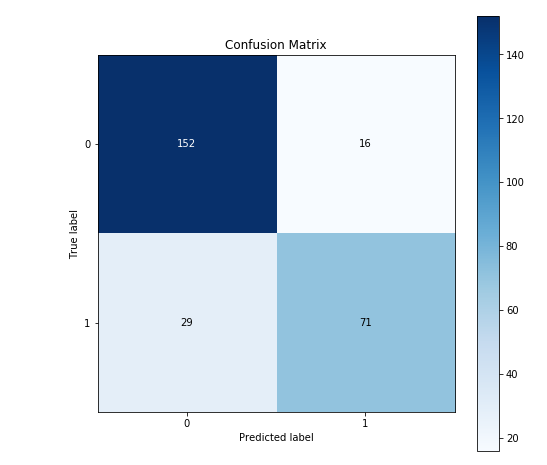
Para conocer los hiperparámetros encontrados en esta versión del algoritmo utilizamos:
print("Best: %f using %s" % (grid_win.best_score_, grid_win.best_params_))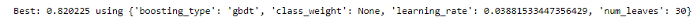
Fila 4: Aplicación de hiperoptimización bayesiana con la biblioteca Hyperopt.
Debo confesar que antes de entender cómo aplicar la técnica de optimización bayesiana, me sentí bastante perdido y confundido, hasta el punto de estar cerca de rendirme.
La documentación y los ejemplos tanto de la biblioteca como de otros artículos que podría tomar como referencias son bastante vagos, demasiado aburridos o muy desactualizados.
Todo esto hasta que encontré este excelente tutorial⁵ y su código ⁶ de Will Koehrsen , el cual aconsejo revisar y probar paso a paso ya que es claro y exhaustivo. Siguiéndolo a él, lo primero que vamos a hacer es definir nuestra función objetivo que debe devolver un diccionario al menos con las etiquetas 'loss' y 'status'.
import csv
from hyperopt import STATUS_OK
from timeit import default_timer as timer
MAX_EVALS = 500
N_FOLDS = 10
def objective(params, n_folds = N_FOLDS):
"""Función objetivo para la Optimización de hiperparametros del Gradient Boosting Machine"""
# Llevar el conteo de iteraciones
global ITERATION
ITERATION += 1
# Recupera el subsample si se encuentra, en caso contrario se asigna 1.0
subsample = params['boosting_type'].get('subsample', 1.0)
# Extrae el boosting type
params['boosting_type'] = params['boosting_type']
['boosting_type']
params['subsample'] = subsample
# Se asegura que los parametros que tienen que ser enteros sean enteros
for parameter_name in ['num_leaves', 'subsample_for_bin',
'min_child_samples']:
params[parameter_name] = int(params[parameter_name])
start = timer()
# realiza n_folds de cross validation
cv_results = lgb.cv(params, train_set, num_boost_round = 10000,
nfold = n_folds, early_stopping_rounds = 100,
metrics = 'auc', seed = 50)
run_time = timer() - start
# Extrae el mejor score
best_score = np.max(cv_results['auc-mean'])
# El loss se debe minimizar
loss = 1 - best_score
# Impulsando las iteraciones que arrojaron el mayor score en CV
n_estimators = int(np.argmax(cv_results['auc-mean']) + 1)
# Escribe sobre el archivo CSV ('a' significa append)
of_connection = open(out_file, 'a')
writer = csv.writer(of_connection)
writer.writerow([loss, params, ITERATION, n_estimators,
run_time])
# Dictionary con informacion para la evaluación
return {'loss': loss, 'params': params, 'iteration': ITERATION,
'estimators': n_estimators, 'train_time': run_time,
'status': STATUS_OK}“El Espacio del Dominio: El Dominio representa el rango de valores que queremos evaluar para cada hiperparámetro. En cada iteración de la búsqueda, el algoritmo de optimización bayesiano elegirá un valor para cada hiperparámetro desde el espacio del domino. Cuando hacemos un Random Search o un Grid Search, el espacio del dominio es una cuadrícula (una tabla de valores establecidos). En la optimización bayesiana, la idea es la misma, excepto que este espacio tiene distribuciones de probabilidad para cada hiperparámetro en lugar de valores discretos.”
space = {
'class_weight': hp.choice('class_weight', [None, 'balanced']),
'boosting_type': hp.choice('boosting_type', [
{'boosting_type': 'gbdt', 'subsample': hp.uniform('gdbt_subsample', 0.5, 1)},
{'boosting_type': 'dart', 'subsample': hp.uniform('dart_subsample', 0.5, 1)},
{'boosting_type': 'goss', 'subsample': 1.0}]),
'num_leaves': hp.quniform('num_leaves', 30, 150, 1),
'learning_rate': hp.loguniform('learning_rate', np.log(0.01),np.log(0.2)),
'subsample_for_bin': hp.quniform('subsample_for_bin', 20000,300000),
'min_child_samples': hp.quniform('min_child_samples', 20, 500, 5),
'reg_alpha': hp.uniform('reg_alpha', 0.0, 1.0),
'reg_lambda': hp.uniform('reg_lambda', 0.0, 1.0),
'colsample_bytree': hp.uniform('colsample_by_tree', 0.6, 1.0)}Aquí se pueden usar diferentes tipos de distribución de dominio (se puede conseguir la lista completa de distribuciones en la documentación de hyperopt):
- choice : variables categóricas
- quniform : discretas uniformes (números enteros espaciados uniformemente)
- uniform: continuad uniformes (floats espaciados uniformemente)
- loguniform: logarítmicas continuas uniformes (floats espaciados uniformemente en una escala logaritmica)
from hyperopt import tpe
from hyperopt import Trials
# Algoritmo de optimización
tpe_algorithm = tpe.suggest
# Lleva el registro de los resultados
bayes_trials = Trials()
# archivo para guardar los primeros resultados
out_file = './gbm_trials.csv'
of_connection = open(out_file, 'w')
writer = csv.writer(of_connection)
# escribe la cabecera de los archivos
writer.writerow(['loss', 'params', 'iteration', 'estimators', 'train_time'])
of_connection.close()Finalmente, con todo el código listo, buscamos la mejor combinación de parámetros a través de la función fmin:
from hyperopt import fmin
# Variable Global
global ITERATION
ITERATION = 0
MAX_EVALS = 100
# Crea un dataset lgb
train_set = lgb.Dataset(X_train, label = y_train)
# Corre la optimización
best = fmin(fn = objective, space = space, algo = tpe.suggest,
max_evals = MAX_EVALS, trials = bayes_trials,
rstate =np.random.RandomState(50))Esta función activa el proceso de búsqueda de la mejor combinación. En el ejemplo original, la variable MAX_EVALS se estableció en 500; debido a problemas de rendimiento para este ejercicio, se redujo a 100, lo que puede haber afectado el resultado final.

Una vez finalizado el proceso, podemos tomar el objeto Trials (bayes_trials en nuestro caso) y analizar sus resultados:
# Ordena las pruebas segun el menor loss (mayor AUC) primero
bayes_trials_results = sorted(bayes_trials.results, key = lambda x: x['loss'])
bayes_trials_results[:2]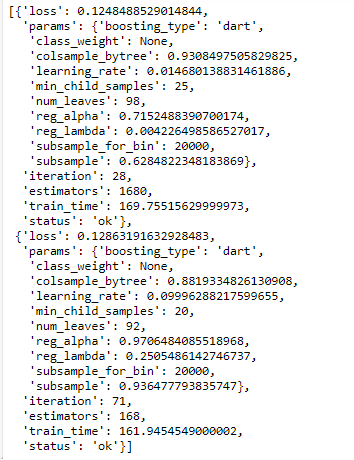
También podemos cargar Dataframe desde el CSV y usar la libreria 'ast' para transformar un texto en un diccionario y alimentar nuestro modelo final con el mejor resultado.
results = pd.read_csv('./gbm_trials.csv')
# Ordena con el mejor score de primero y resetea el indice para las divisiones
results.sort_values('loss', ascending = True, inplace = True)
results.reset_index(inplace = True, drop = True)
results.head()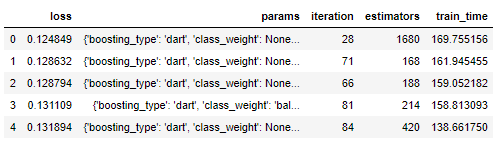
import ast
# Convierte de string a un dictionary
ast.literal_eval(results.loc[0, 'params'])
# Extrae el numero ideal de Estimators e hiperparametros
best_bayes_estimators = int(results.loc[0, 'estimators'])
best_bayes_params = ast.literal_eval(results.loc[0, 'params']).copy()
# Recrea el mejor modelo y lo entrena con el set de training
best_bayes_model = lgb.LGBMClassifier(n_estimators=best_bayes_estimators, n_jobs = -1,
objective = 'binary', random_state = 7,
**best_bayes_params)
best_bayes_model.fit(X_train, y_train)
# Evalua sobre el set de testing
preds = best_bayes_model.predict(X_valid)
print(confusion_matrix(y_valid, preds))
print (best_bayes_model.score(X_valid,y_valid))
skplt.metrics.plot_confusion_matrix(y_valid, preds,figsize=(8,8))
resul.iloc[3,7] = best_bayes_model.score(X_valid,y_valid)
resul.head()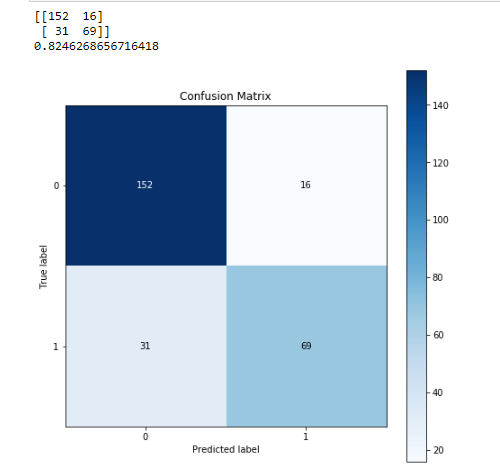
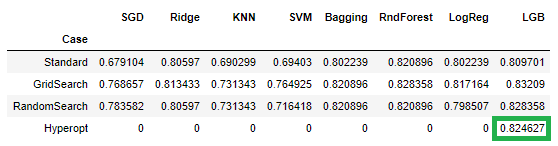
Tarea para el lector! El post ya quedó largo para adecuar el hyperopt a los restantes algoritmos, los/as invitamos a adecuar el script a estos modelos.
Resumen
Hemos desarrollado el código completo para aplicar tres técnicas de optimización disponibles, sobre ocho algoritmos de clasificación diferentes.
El tiempo y la capacidad computacional necesarios para aplicar estas técnicas es un tema a tener en cuenta; es recomendable y necesario aplicarlas cuando el modelo seleccionado ya está suficientemente probado y bien construido como para que valga la pena los recursos empleados.
Se debe tener en cuenta también que la optimización de parámetros no implica necesariamente una mejora continua en los resultados del modelo aplicado sobre los datos de prueba, ya que la selección de parámetros podría estar generando un sobreajuste (overfitting).
Finalmente, cabe destacar que las mejoras observadas en los diferentes modelos han sido en general de una magnitud considerable, lo que deja al menos la puerta abierta a la posibilidad de incrementar el rendimiento del algoritmo a través de alguna de estas técnicas.
Fuentes y Referencias
[1] https://en.wikipedia.org/w/index.php?title=Accuracy_and_precision&action=edit§ion=4
En calsificacion binaria el Accuracy tambien es usado como una medida estadística sobre que tan bien se identifica o excluye una condición en una prueba del modelo. Es decir, el Accuracy es la proporción de resultados verdaderos (tanto Positivos verdaderos como Negativos verdaderos) entre el total de casos examinados.. La formula para calcular el Accuracy es:
Accuracy = (TP+TN)/(TP+TN+FP+FN)
Donde: TP = True positive (Positivo verdadero); FP = False positive(Positivo falso); TN = True negative(Negativo verdadero); FN = False negative(Negativo falso)
[2] https://machinelearningmastery.com/hyperparameters-for-classification-machine-learning-algorithms/
[4] https://medium.com/apprentice-journal/hyper-parameter-optimization-c9b78372447b
Adaptación y traducción por Luis Andres Marquez Vergara, github.
Artículo original en inglés por Gabriel Naya:
Publicado originalmente en español en escueladedatosvivos.ai
¿Te interesa seguir aprendiendo?



